|
近日,中国农业科学院深圳农业基因组研究所刘毓文课题组与广西大学赵云翔课题组合作,在基因组育种共性技术领域取得了重要创新性突破。相关研究成果以《DPImpute: A Genotype Imputation Framework for Ultra-low Coverage Whole-genome Sequencing and its Application in Genomic Selection》为题,于2月27日在线发表于国际知名期刊Advanced Science。该研究开发了一种全新的基因型填充计算框架DPImpute,为小样本量超低覆盖度全基因组测序(ulcWGS)提供了高效、精准的解决方案,显著降低了基因组选择的成本,提高了应用场景的灵活性,并首次实现了动物囊胚(杜洛克猪)单细胞全基因组SNP基因型准确分型,为胚胎基因组选择提供了重要的技术支撑。
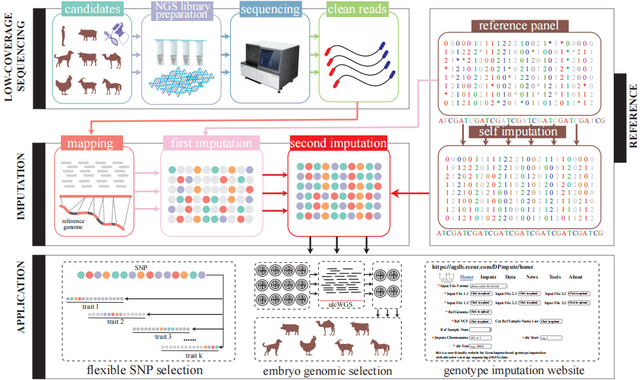
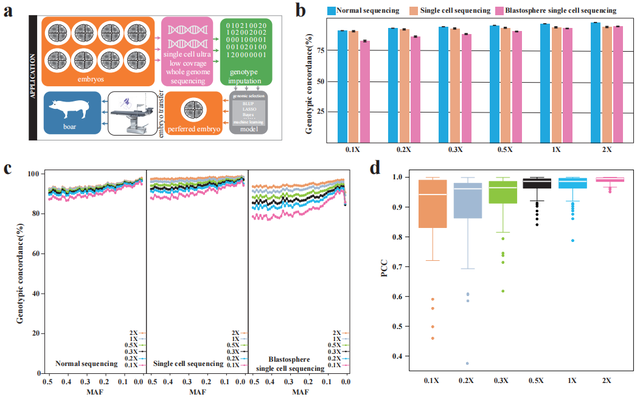
研究背景与挑战 全基因组测序(WGS)在揭示基因型与表型之间复杂关系的过程中发挥着至关重要的作用,尤其在精准表型预测和基因组选择(GS)等领域提供了丰富的基因组信息。然而,尽管WGS为基因组学研究提供了前所未有的分辨率和全面性,其在育种领域的广泛应用仍面临高测序深度带来的成本和计算压力。虽然基于基因型填充的策略可以降低测序深度,但在超低覆盖度( 研究创新与成果 DPImpute平台采用了一种全新的两步基因型填充方法,能够在超低覆盖度全基因组测序(ulcWGS)条件下,显著提高基因型填充的准确性。该平台首次实现了对单个动物囊胚细胞(来源于杜洛克猪囊胚)进行千万级别全基因组SNP的准确分型,并直接用于预测对应囊胚的经济性状(包括瘦肉率、背膘厚度、眼肌面积等),获得了良好的预测稳定性。这一技术体系不仅大大降低了测序成本,还为动物胚胎复杂性状表型的预测和胚胎基因组选择提供了重要的技术支撑。 技术原理与优势 DPImpute采用两轮填充策略,旨在实现高精度的SNP填充(图1)。在第一轮填充中,利用高准确度测序样本的参考群体基因型数据,结合GLIMPSE分析工具,构建单倍型并填充测试样本的SNP。在第二轮填充中,通过Beagle对参考面板进行自我填充,进一步优化基因型信息。这一迭代策略有效解决了超低覆盖度测序数据中因测序随机性和不完整遗传信息带来的挑战,显著提升了填充的准确性和稳健性。
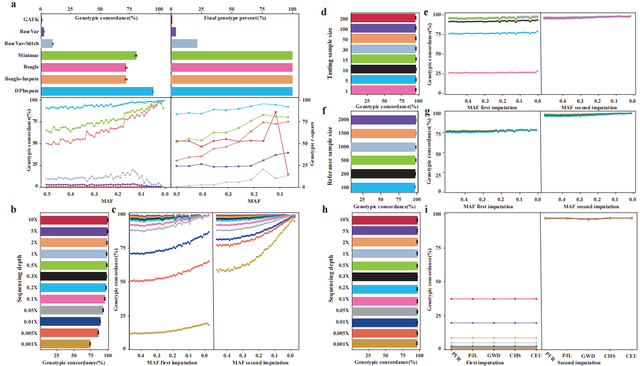
图1.DPImpute框架的示意图 性能评估与验证 研究团队通过多组实验验证了DPImpute的优越性能。在参考面板较小(≤100)、测试样本数较少(≤10)以及超低覆盖度测序(≤0.1X)等挑战性条件下,DPImpute的平均基因型一致性准确度达到94.80%,显著优于现有方法(图2)。单个样本超低覆盖度(0.3X),有参考群情况下,也有较高(98.29%)的准确率(图2)。此外,DPImpute在不同测序深度、测试样本大小、参考群体大小以及跨种群条件下均表现出高准确性和稳定性,展现了其广泛的应用潜力。
图2.参考样本大小下的填充性能比较 应用前景与意义 DPImpute不仅为动物育种提供了低成本、高精度的基因分型解决方案,还为人类胚胎植入前遗传检测(PGT)提供了新的技术路径(图3)。研究团队开发的用户友好型网络平台(https://agdb.ecenr.com/DPImpute/home)和Docker容器,进一步降低了DPImpute的使用门槛,为科研人员提供了便捷的工具支持。
图3.单细胞基因型填充 研究团队与资助 刘毓文课题组博士生郑伟刚(华中农业大学联合培养博士)和马文龙博士为本文的共同第一作者;中国农业科学院深圳基因组所刘毓文研究员和广西大学赵云翔教授为本研究的共同通讯作者。唐中林研究员和李奎教授提供了样本资源的帮助。研究得到了国家自然科学基金项目、国家重点研究与发展计划项目和十四五重点研发项目的支持。 DPImpute平台通过精确且成本效益高的基因分型,显著加速了育种计划的进程,成为人类与动物群体基因分型研究中的一项宝贵工具。该研究不仅为基因组选择提供了新的技术路径,还为胚胎全基因组测序与复杂性状评估开辟了全新的研究方向,具有重要的科学意义和应用价值。 |